大模型API服务(scRNA自动注释)#
[!NOTE]
本服务旨在提供GPT的API服务,可以用于各种支持API的软件,用户能够在不挂梯子的前提下,使用个人API和KEY进行编程调用(批量总结几千篇pdf文档,单细胞自动注释等任务)。
1.在淘宝购买需要的API,客服会给你兑换码
淘宝店铺链接:【淘宝】https://e.tb.cn/h.htKrXhJveLJwJKM?tk=C4BF49bPM2D CZ193 「数信院生信AI助手scRNA注释AI神器独享web端使用神器」 点击链接直接打开 或者 淘宝搜索直接打开
2.使用手机/平板/电脑等设备,进入数信云API服务平台
打开浏览器进入网址:https://api.sxycloud.comd
新用户首次使用需要注册账号(点击右上角Sign in)

3.登录成功之后可以进入“操练场”,分组选择“操练场对话专用”,然后可以选择GPT5/Claude/DeepSeek等热门模型

4.点击“钱包管理”栏,输入客服给的兑换码进行额度兑换
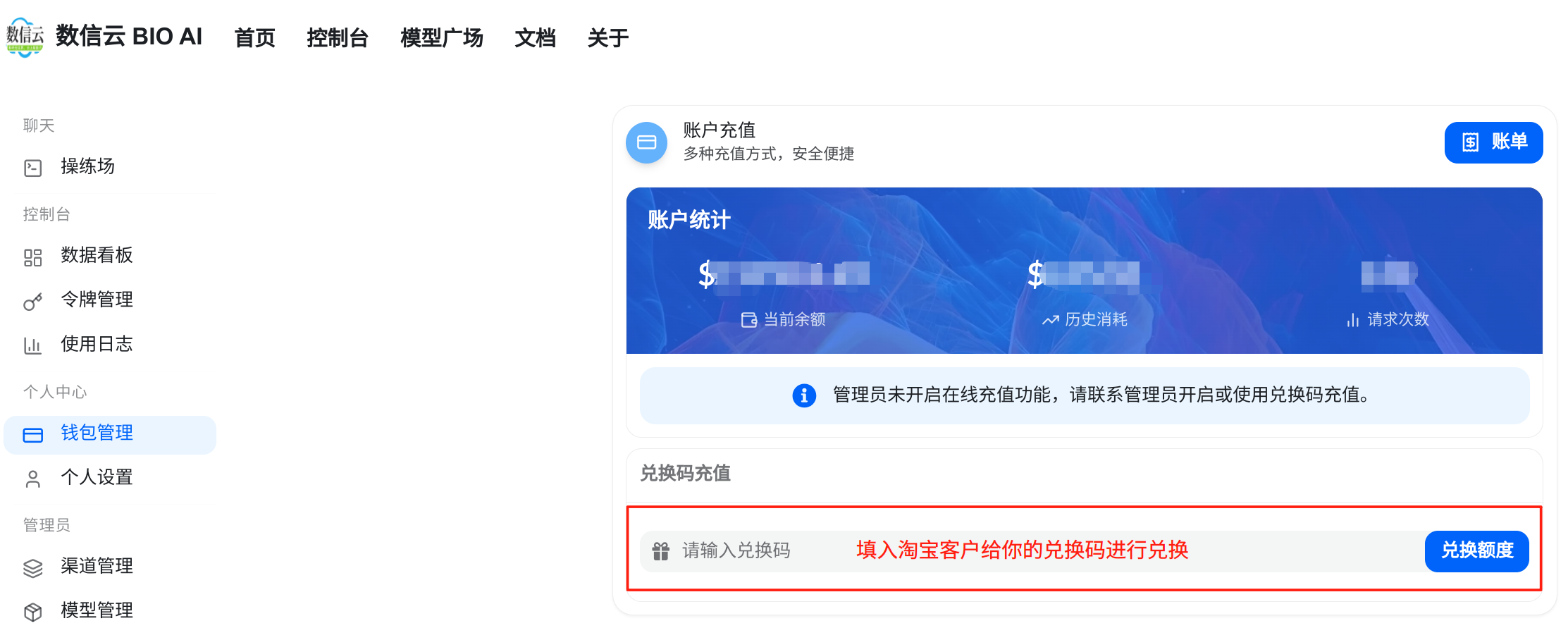
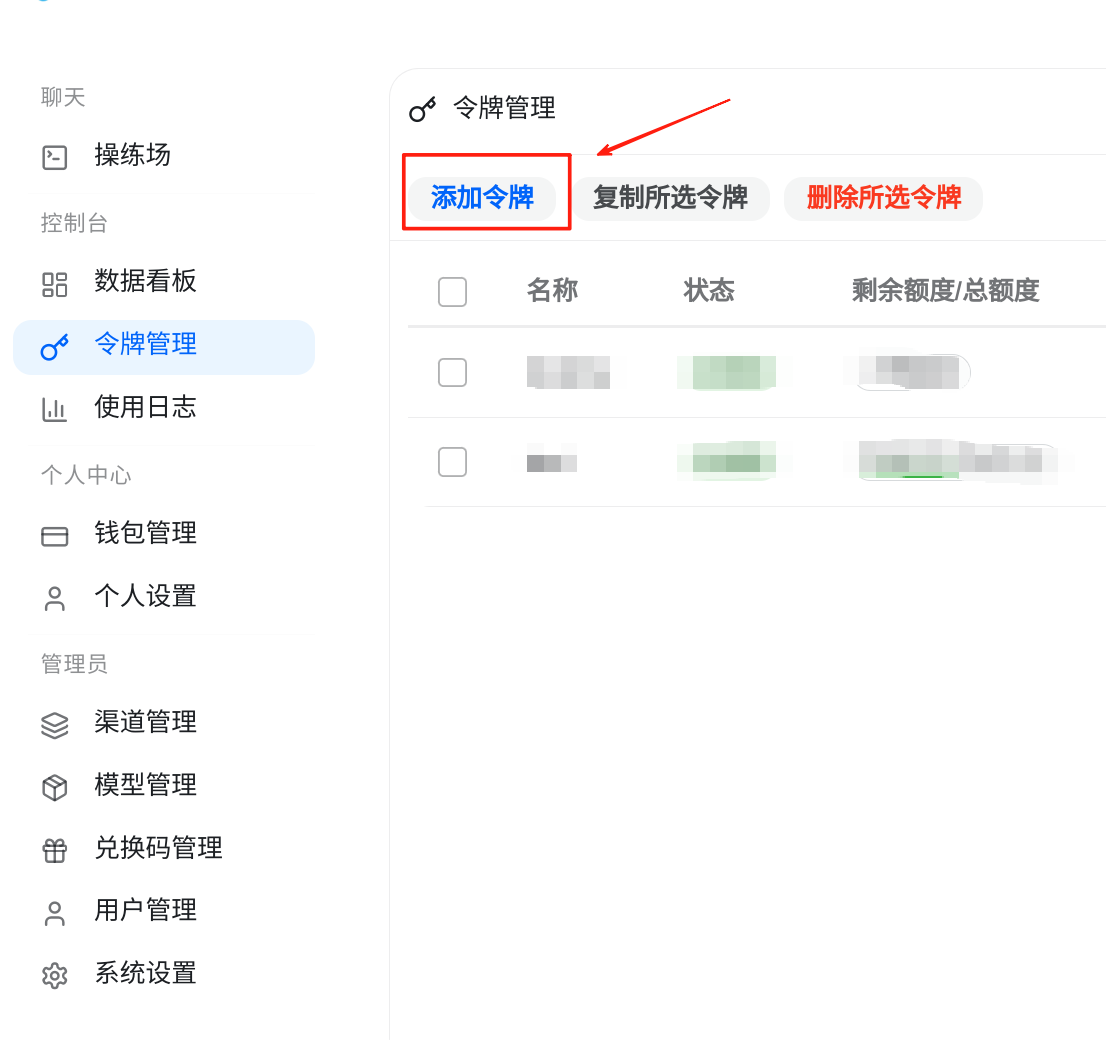
5.点击“令牌管理”栏,再点击“添加令牌”,即可得到你的API-KEY
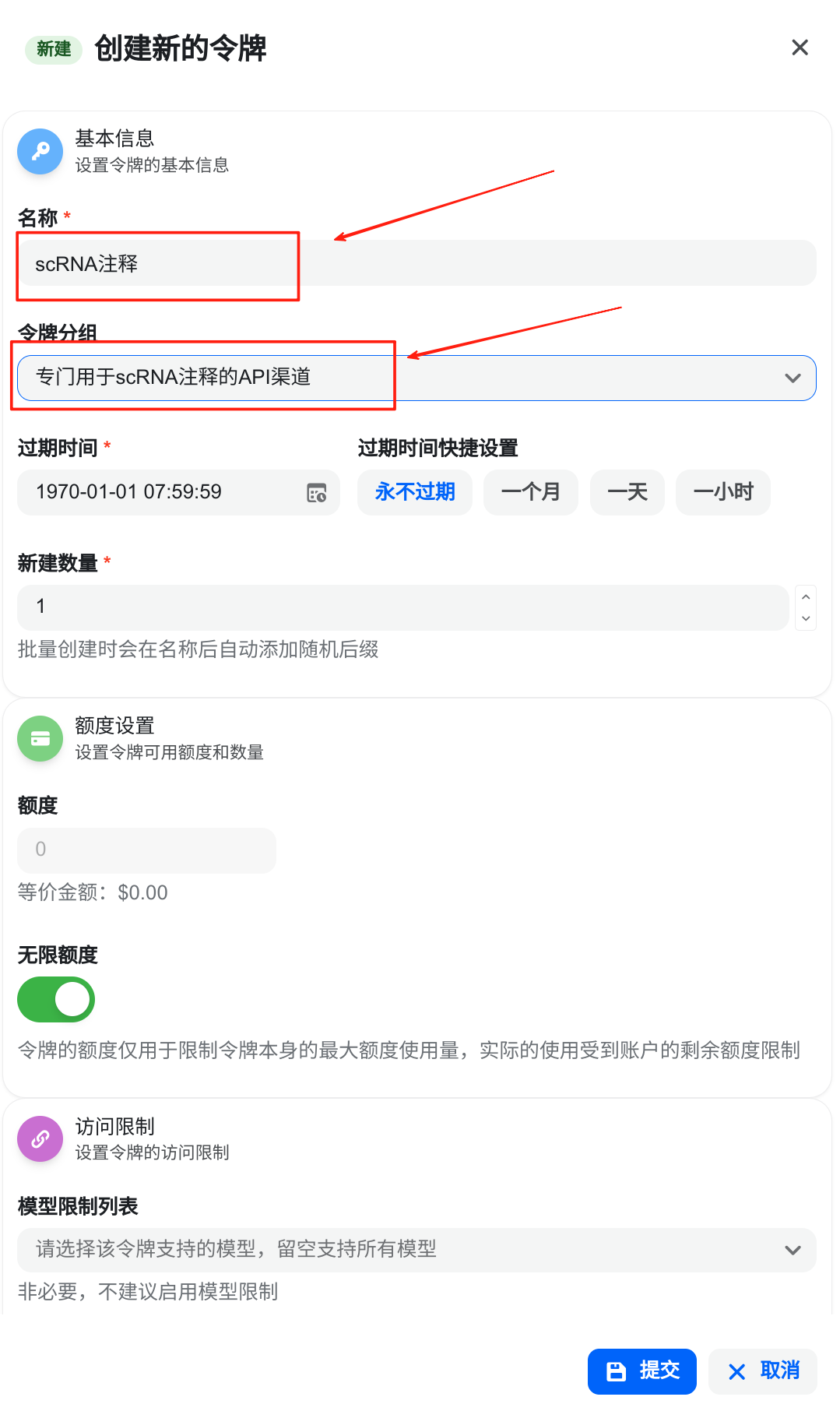
然后“创建新的令牌”,这里的”令牌分组“一定要选”专门用于scRNA注释的API渠道“
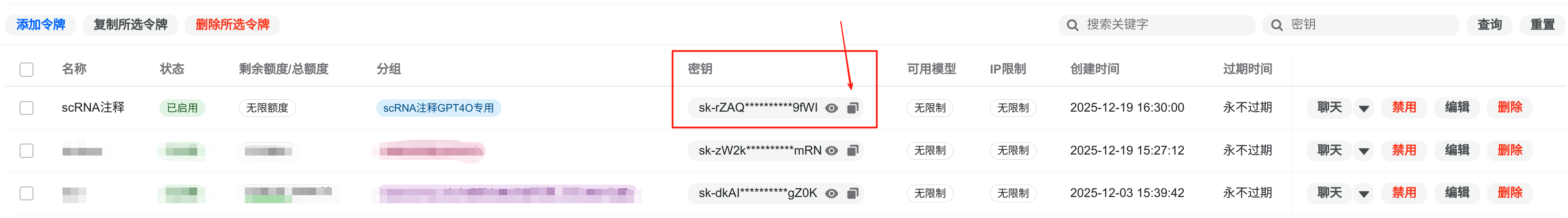
最后我们点击“复制”图标即可得到API-KEY:sk-xxxxxxxxx
6.单细胞注释工具——GPTcelltype(发表在Nature Method)
具体教程请查看https://github.com/EddieLv/GPTCelltype
基于GPT4模型对scRNA分群进行自动注释(国内网络可用)
安装数信院GPT注释的两个包
install.packages("remotes")
remotes::install_github("EddieLv/apiSXY", force=T)
remotes::install_github("EddieLv/GPTCelltypeSXY", force=T)
导入安装的R包,并设置GPT令牌
# Load packages
library(GPTCelltypeSXY)
library(apiSXY)
# 'sk-XXXX'是令牌,可以找数信院客服付费领取(或淘宝搜索'数信院生信服务器'购买)
Sys.setenv(OPENAI_API_KEY = 'sk-XXXX')
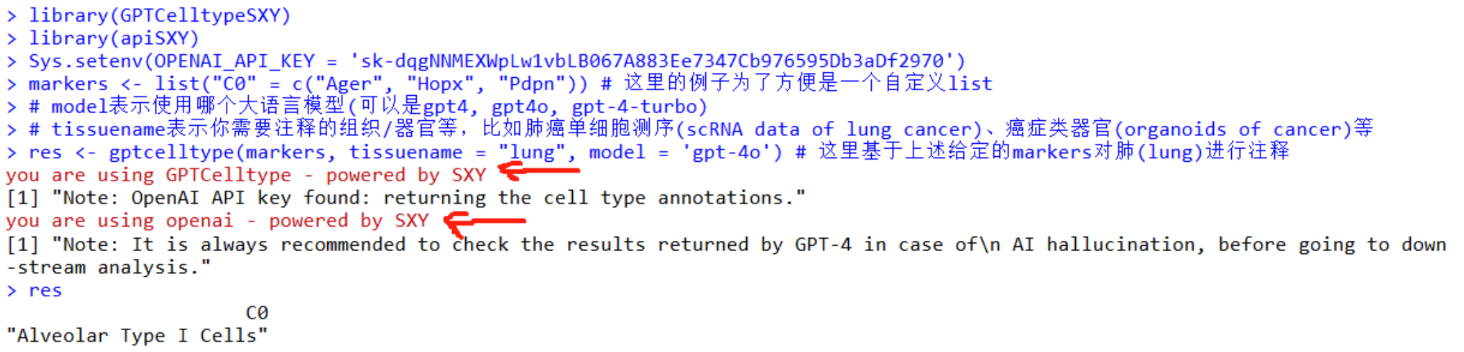
使用GPT进行自动注释
# markers是一个list, 也可以是Seurat FindAllMarkers()出来的data.frame
markers <- list("C0" = c("Ager", "Hopx", "Pdpn")) # 这里的例子为了方便是一个自定义list
# model表示使用哪个大语言模型(可以是gpt4, gpt4o, gpt-4-turbo)
# tissuename表示你需要注释的组织/器官等,比如肺癌单细胞测序(scRNA data of lung cancer)、癌症类器官(organoids of cancer)等
res <- gptcelltype(markers, tissuename = "lung", model = 'gpt-4o') # 这里基于上述给定的markers对肺(lung)进行注释
res
示例过程(注意2处红线指出的地方,代表成功调用了函数)
关于 GPTCelltypeSXY 包,这里有几个使用时的注意事项:
1.基于标记基因(Markers)注释 GPTCelltypeSXY 包是基于标记基因(Markers)来进行细胞类型注释的。
因此选择标记基因时要小心,不要提供过于冗余或重复的标记基因(特别是FindAllMarkers的结果要先进行人工过滤再投入gptcelltype模型)。冗余的标记基因可能会导致模型过拟合,影响注释的准确性。
确保标记基因的可靠性和特异性,应当尽量选择已经在文献中验证过的标记基因,并且能够明确区分不同细胞类型的基因。
精简标记基因集,提供少量但高特异性的标记基因集,能提高模型的性能,避免信息过载。
2.细胞类型库的选择 GPTCelltypeSXY 包基于特定的细胞类型数据库进行注释。
使用时要确保选择适合你的数据集的细胞类型库。例如,不同物种、组织或实验条件下,可能需要不同的标注系统。确保数据集中的细胞类型与所使用的库相匹配,以避免误注释。
